
Uma equipa internacional desenvolveu a árvore genealógica de aves mais abrangente, cobrindo 93 milhões de anos e 92% das famílias de aves, utilizando métodos computacionais avançados e recursos de supercomputação. Crédito: SciTechDaily.com
Uma equipe global de pesquisadores construiu a árvore genealógica de aves mais abrangente e extensa até hoje, detalhando as conexões evolutivas entre 363 aves. espécies mais de 93 milhões de anos. Este gráfico representa 92% de todas as famílias de aves.
O avanço foi possível em grande parte graças aos métodos computacionais de ponta desenvolvidos por engenheiros da Universidade da Califórnia em San Diego, combinados com os recursos de supercomputação de última geração da universidade no San Diego Supercomputer Center. Essas tecnologias permitiram aos pesquisadores analisar grandes quantidades de dados genômicos com alta precisão e velocidade, lançando as bases para a construção da árvore genealógica de aves mais completa já montada.
O avanço está detalhado em dois artigos complementares publicados em 1º de abril no Natureza e a Anais da Academia Nacional de Ciências (PNAS). A árvore genealógica atualizada, relatada em Naturezarevelou padrões na história evolutiva das aves após o evento cataclísmico de extinção em massa que exterminou os dinossauros há 66 milhões de anos.
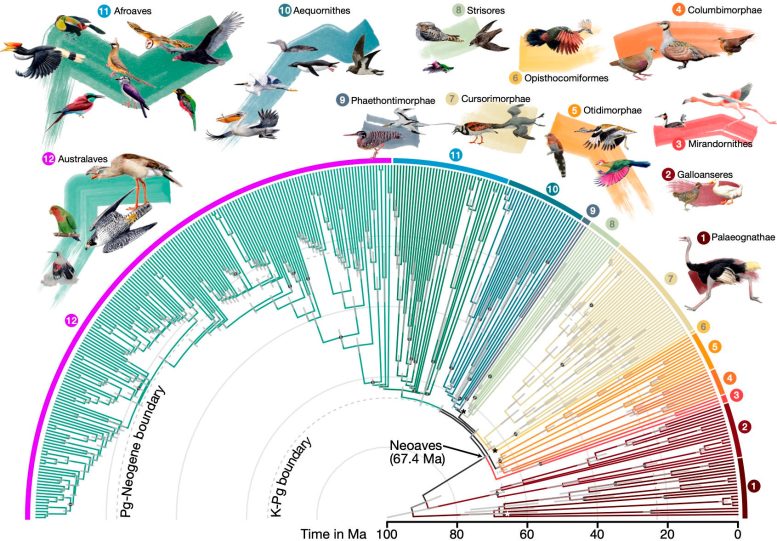
A árvore genealógica atualizada das aves, publicada em Natureza, delineando 93 milhões de anos de relações evolutivas entre 363 espécies de aves. Crédito: Jon Fjeldså (desenhos) e Josefin Stiller
Os investigadores observaram aumentos acentuados no tamanho efectivo da população, nas taxas de substituição e no tamanho relativo do cérebro nas primeiras aves, lançando uma nova luz sobre os mecanismos adaptativos que impulsionaram a diversificação aviária no rescaldo deste evento crucial. No artigo complementar publicado em PNASos investigadores examinaram de perto um dos ramos da nova árvore genealógica e descobriram que os flamingos e as pombas estão mais distantemente relacionados do que as análises anteriores do genoma tinham mostrado.
O trabalho faz parte Projeto Bird 10.000 Genomas (B10K)um esforço multiinstitucional liderado pela Universidade de Copenhague, Universidade de Zhejiang e UC San Diego que visa gerar rascunhos de sequências genômicas para cerca de 10.500 espécies de aves existentes.
“Nosso objetivo é reconstruir toda a história evolutiva de todas as aves”, disse Siavash Mirarab, professor de engenharia elétrica e de computação na Escola de Engenharia Jacobs da UC San Diego, que é co-autor sênior do Natureza artigo, bem como primeiro e co-autor no PNAS papel.
Juntando o passado
No centro desses estudos está um conjunto de algoritmos conhecido como ASTRAL, que o laboratório de Mirarab desenvolveu para inferir relações evolutivas com escalabilidade, precisão e velocidade sem precedentes. Ao aproveitar o poder desses algoritmos, a equipe integrou dados genômicos de mais de 60.000 regiões genômicas, fornecendo uma base estatística robusta para suas análises.
Os pesquisadores examinaram então a história evolutiva de segmentos individuais do genoma. A partir daí, eles montaram um mosaico de árvores genéticas, que foram então compiladas em uma árvore de espécies abrangente. Esta abordagem meticulosa permitiu aos investigadores construir uma nova e melhorada árvore genealógica de aves que delineia eventos ramificados complexos com notável precisão e detalhe, mesmo em casos de incerteza histórica.
“Descobrimos que o nosso método de adicionar dezenas de milhares de genes à nossa análise era realmente necessário para resolver as relações evolutivas entre espécies de aves”, disse Mirarab. “Você realmente precisa de todos esses dados genômicos para recuperar o que aconteceu neste determinado período de tempo, 65-67 milhões de anos atrás, com alta confiança.”

No estudo publicado na PNAS, os pesquisadores examinaram de perto um dos ramos da árvore genealógica atualizada das aves e descobriram que grupos incluindo flamingos e pombas são mais distantemente relacionados do que as análises anteriores do genoma haviam mostrado e atribuíram o resultado a uma região incomum do cromossomo. 4. Crédito: Ed Braun (desenhos), Daniel J. Field (imagens de pássaros) e Siavash Miarab
A capacidade da equipe de conduzir essas análises em enormes conjuntos de dados foi possível porque o laboratório de Mirarab projetou seus métodos computacionais para rodar em poderosas máquinas GPU. Eles fizeram seus cálculos no Extensão supercomputador no Supercomputador de San Diego na UC San Diego.
“Tivemos a sorte de ter acesso a um supercomputador de última geração”, disse Mirarab. “Sem Extensãonão teríamos sido capazes de executar e reexecutar nossas análises em conjuntos de dados tão grandes em um período de tempo razoável.”
Os pesquisadores também analisaram os efeitos de diferentes métodos de amostragem do genoma na precisão da árvore. Eles mostraram que duas estratégias – sequenciar muitos genes de cada espécie, bem como sequenciar muitas espécies – combinadas são importantes para reconstruir esta história evolutiva.
“Como usamos uma mistura de ambas as estratégias, pudemos testar qual abordagem tem impactos mais fortes na reconstrução filogenética”, disse Josefin Stiller, professor de biologia na Universidade de Copenhague e principal autor do estudo. Natureza papel. “Descobrimos que era mais importante amostrar muitas sequências genéticas de cada organismo do que amostrar uma gama mais ampla de espécies, embora o último método nos tenha ajudado a datar quando diferentes grupos evoluíram.”
Corrigindo o passado
Com a ajuda dos seus métodos computacionais avançados, os investigadores também conseguiram esclarecer algo invulgar que tinham descoberto num dos seus estudos anteriores: uma secção específica de um cromossoma no genoma da ave permaneceu inalterada durante milhões de anos, anulada. dos padrões esperados de recombinação genética.
Esta anomalia inicialmente levou os pesquisadores a agrupar incorretamente flamingos e pombas como primos evolutivos, pois eles pareciam intimamente relacionados com base nesta seção inalterada de ADN. Isso porque a análise anterior foi baseada nos genomas de 48 espécies de aves. Mas, ao repetir a análise usando os genomas de 363 espécies, surgiu uma árvore genealógica mais precisa que afastou os pombos dos flamingos. Além disso, usando seis genomas de alta qualidade fornecidos pelo Projeto Genoma de Vertebrados (VGP) – liderado pelo coautor Erich Jarvis, professor de neurobiologia na Universidade Rockefeller – Mirarab e colegas conseguiram detectar e explicar supostamente este padrão surpreendente.
“O que é surpreendente é que este período de recombinação suprimida pode induzir a análise em erro”, disse Edward Braun, professor de biologia no Universidade da Flórida e co-autor correspondente do PNAS papel. “E porque poderia enganar a análise, foi na verdade detectável mais de 60 milhões de anos no futuro. Essa é a parte legal.”
Próximos passos
O impacto deste trabalho vai muito além do estudo da história evolutiva das aves. Os métodos computacionais pioneiros do laboratório de Mirarab tornaram-se uma das ferramentas padrão para reconstruir árvores evolutivas para uma variedade de outros animais.
Seguindo em frente, a equipe continua seus esforços para construir um quadro completo da evolução das aves. Os biólogos estão trabalhando no sequenciamento dos genomas de outras espécies de aves na esperança de expandir a árvore genealógica para incluir milhares de gêneros de aves. Entretanto, os cientistas computacionais liderados por Mirarab estão a refinar os seus algoritmos para acomodar conjuntos de dados ainda maiores, a fim de garantir que as análises em estudos futuros sejam conduzidas com alta velocidade e precisão.
Referência: “Complexidade da evolução aviária revelada pelos genomas de nível familiar” por Josephine Stiller, Shaohong Feng, Al-Aabid Chowdhury, Iker Rivas-Gonzalez, David A. Duchene, Qi Fang, Yuan Deng, Alexey Kozlov, Alexandros Stamatakis, James Claramunt , Jacqueline MT Nguyen , Simon YW Ho , Brant C. Faircloth , Julia Haag , Peter Houde , Joel Cracraft , Metin Balaban , Uyen Mai , Guangji Chen , Rongsheng Gao , Chengran Zhou , Yulong Xie , Zijian Huang , Zhen Cao , Zhi Yan , Huw A. Ogilvie, Luay Nakhleh, Bent Lindow, Benoit Morel, Jon Fjeldse, Peter A. Hosner, Rute R. da Fonseca, Bent Petersen, Joseph A. Tobias, Tamas Szekely, Jonathan David Kennedy, Andrew Hart Reeve, Andras Liker, Martin Starvander, Agostinho Antunes, Dieter Thomas Tietze, Mads Bertelsen, Fumin Lei, Carsten Rahbek, Gary R. Graves, Mikkel H. Schierup, Tandy Warnow, Edward L. Braun, M. Thomas P. Gilbert, Erich D. Jarvis, Siavash Mirarab e Guojie Zhang, 1º de abril de 2024, Natureza.
DOI: 10.1038/s41586-024-07323-1
“Uma região de recombinação suprimida engana a filogenômica neoaviana” por Siavash Mirarab, Iker Rivas-Gonzalez, Shaohong Feng, Josephine Stiller, Qi Fang, Uyen Mai, Glenn Hickey, Guangji Chen, Nadolina Brajuka, Olivier Fedrigo, Giulio Formenti, Jochen BW Wolf, Kerstin Howe, Agostinho Antunes, Mikkel H. Schierup, Benedict Paten, Erich D. Jarvis, Guojie Zhang e Edward L. Braun, 1 de abril de 2024, Anais da Academia Nacional de Ciências.
DOI: 10.1073/pnas.2319506121